在他的畅销书《上帝的语言》(2006)[ 译注:中文版2010]中, 遗传学家柯林斯声称人类DNA提供了"强有力的支持达尔文进化论的论据,即人与其他生物是从一个共同祖先借着自然选择和随机变异的操作进化而来。(注1) 具体地说, 他认为我们的DNA表明, 人类和类人猿有共同的祖先。
柯林斯是前任人类基因组计划的负责人,也是一个众所周知的福音派基督徒,他支持达尔文的进化论和胚胎干细胞的研究。(注2)。借着约翰.邓普顿基金会2008年的二百万美元赠款的帮助,柯林斯与他人共同创立生物与道(BioLogos)基金会,目的是要说服基督教领袖及普通民众来接受生物进化论。(注3)柯林斯被奥巴马总统任命为美国国立卫生研究院主任后辞去BioLogos主持人的职位, 但他的猿/人类有共同的祖先的立场还是在各界信徒中有广泛的影响力。
柯林斯提供了两个主要以DNA为基础的论据,支持人类与猩猩等动物有共同的祖先的立场。首先,人类和其他哺乳动物共有的不编码蛋白质的DNA是假想无功能的垃圾,根据柯林斯的立场, "一个人类和小鼠共同的祖先的结论是几乎不可避免的。"(注4) 其次, 人类的第二号染色体是从像猿的两种染色体融合的结果, 柯林斯声称这些证据的发现是"很难理解的……除非有一个人类和猿共同的祖先之假设。"(注5)
这些都是常见的人类和猿共同的祖先的论据, 但这一章将显示,柯林斯的立场主要是基于过时的科学证据和有问题的假设。具体而言:
o 大量的研究已经发现大量证据支持不编码蛋白质的DNA的功能, 表明它毕竟不是遗传的"垃圾"。
o 人类染色体融合可能意味着人类的血统中经历了一个融合过程, 但是这并没有告诉我们是否我们的血统可以追溯到与猿分享的共同祖先。此外, 支持人类染色体融合的基因证据远不如柯林斯和其他人所说的那样充足。
总之,从DNA而来的证据没有确定柯林斯有关人类进化的结论。
不编码蛋白质的DNA毕竟不是遗传的"垃圾"
柯林斯避免了通常的简单说法: 两个物种之间的共用遗传功能相似性必然证明他们共用一个相同的祖先, 他承认遗传功能的相似性"本身当然并不能证明有一个共同的祖先"。因为一位设计者可以重复地使用"成功的设计原则"。 (注6)。相反地,柯林斯提供一个不同的观点: 他列举了古老重复元素 (Ancient Repetitive Elements: AREs) , 被称为没有功能的垃圾 DNA, 在他看来, 这证实了达尔文的进化论和人/猿有共同的祖先。
在哺乳动物的基因组中常见重复的元素。我们, 猿, 和老鼠都有。它们在我们的基因组中常出现在同样的位置上。柯林斯坚持这些AREs是"遗传上漂流的废物"(genetic flotsam and jetsam)并且是"对认为神从无中创造所有物种的想法有压倒性的挑战"。(注7) 在他看来, "除非人愿意相信神将这些砍了头的AREs元素放在这些精确的位置上来混淆和误导我们, 这些AREs是来自一个人类和小鼠共同祖先的结论几乎是不可避免的"。 (注8) 听起来柯林斯很像无神论达尔文学者理查。道金斯(Dawkins) 他同样写道, "创造论者应该花一些时间认真地猜测造物主何必在基因组中乱扔垃圾……这些串联重复的 DNA垃圾"。(注9) 值得注意的是柯林斯和道金斯在基本上正在使用一个神学论点"上帝不会那样做"作为科学的争辩。他们的神学立论是否健全将留给他人去衡量, 但他们的科学观点已被证据推翻。
科学文献却与柯林斯和道金斯的结论相反, 甚至粗略审查这些文件,都会表明简单地假设重复的和不编码蛋白质的DNA是无用的基因"垃圾",是极端不恰当的。
开明的科学家在柯林斯写他的书很久以前已经理解这一点。2002年,生物学家理查德·斯滕伯格(Richard Sternberg)查阅科学的文献, 发现了AREs有功能的大量证据。他在纽约科学院年鉴(Annals of the New York Academy of Sciences) 写道: AREs现在的功能包括:
" 卫星重复DNA形成高阶层的核结构
" 卫星重复DNA形成着丝粒(centromeres)
" 卫星重复DNA和其他重复元素参与染色质凝聚的过程
" 端粒(telomeric)串联重复序列和散布核长元件(LINE: Long Interspersed Nuclear Elements)元素
" 次端粒核定位/染色质边界元素(Subtelomeric nuclear positioning)
" 非转座因子(TE: Transposable Elements)穿插染色质的边界元素
" 散布核短元件(SINE: Short Interspersed Nuclear Elements)或重复序列为甲基化核成中心(nucleation centers for methylation)
" 散布核短元件作为染色质边界/绝缘子元素
" 参与细胞增殖的散布核短元件
" 参与细胞应激反应的散布核短元件
" 参与基因表现的散布核短元件(可连接到应激反应)
" 参与结合黏合染色体的散布核短元件
" 参与DNA修复的散布核长元件(注10)
斯滕伯格认为,"自私的垃圾DNA和所有相关框架的叙述必须加入其他新达尔文的进化论"圣像"行列, 尽管他们与实证有差异, 却继续在科学文献中出现。"(注11) 其他遗传研究继续发现各种类型重复DNA的功能,包括散布核短元件(注12),散布核长元件(注13), 和Alu序列(注14), 一份研究报告甚至建议重复的Alu序列可能参与人体中的"高级脑功能的发展"。(注15)许多各类不编码蛋白质的DNA的其他功能已被发现, 其中包括:
" 修复DNA(注16)
" 协助DNA复制(注17)
" 调节DNA转录(注18)
" 协助染色体的折叠和维护(注19)
" 控制RNA编辑和剪接(注20)
" 帮助对抗疾病(注21)
" 调节胚胎发育(注22)
斯滕伯格,以及芝加哥大学遗传学家詹姆斯·夏皮罗在2005年曾预言说:"有一天,我们会认为以前所谓的垃圾DNA?, 乃是真正细胞控制专家系统的重要组成部分。" (注23)
斯滕伯格和夏皮罗所预见的这一天, 可能比预期更快到来。 2007年, 《华盛顿邮报》报道,一个巨大的科学联合项目ENCODE,发现"人类遗传密码的三十亿字母",绝大多数在以前看不见的任务组中忙于劳作"。(注24)根据在《自然》 (Nature)杂志一篇关于该项目的报告:
在不编码蛋白质的DNA被生物学普遍称为"垃圾"DNA的世界里, 我们有一个迷人的但令人迷惑的新认识。一个国际合作研究项目称为DNA元素百科全书(ENCODE)的学者表示, 在含有很少量制造蛋白质的编码序列的选定基因组部分, 介于74%和93%的DNA被转录成核醣核酸RNA。多量不编码蛋白质的DNA具有调节作用, 不同品种的小RNA似乎用以控制基因的表达, 在DNA和RNA转录方式的研究上仅在渐露端倪的开端。(注25)
2007年在《自然遗传学评论》(Nature Reviews of Genetics)名为"全基因组的转录和基因组结构的影响"的论文中,对这些不编码蛋白质的DNA的神秘的广泛复杂功能和重要性质作如下的解释:
证据表明,大多数的人类基因组DNA的两条链都可能被转录,这意味着基因组的转录单位和调节元件的大量重叠。这些观察结果表明,基因组结构不是只有一个功能的,而是代替交织和模块化,并且该相同的基因组序列具有多项功能:即用作多个独立调节的转录物和作为调节区。(注26)
同样地,一篇《科学》 (Science)杂志2008年的报告发现, 几乎所有被详细研究的真核细胞基因组都被转录, 产生巨量不制造蛋白质的RNA, 其中很有可能是有功能的:
科学在过去的几年中已经表明, 所有被研究的真核细胞的基因组几乎都完全被转录,产生巨量的非制造蛋白质的RNA(非编码RNA )。与此同时,人们越来越明显了解,许多这些RNA都有调控功能。在这里我们强调最新的研究进展,说明非编码RNA 控制的基因组动态,细胞生物学和多样性的发展模范。 (注 27)
这报告接着具体地阐述重复元素如何发挥着细胞控制中的功能:"由于大量的重复序列被转录,这可能代表整个基因组控制染色质结构的策略, 并可能被保守贯穿真核生物界"。 (注28)
在2003年的一篇《科学》杂志一篇文章中作者承认, 类似柯林斯所使用的"垃圾DNA "的标签, 实在阻碍了科学家寻索非编码重复元素的功能:
虽然朗朗上口,术语"垃圾DNA "多年来拦阻了主流的科硏人员研究非编码DNA,除了一小部分的基因组的"流浪汉"(clochard)以外, 谁想在基因组的垃圾堆中捡破烂?然而像平时一样, 在科学界也有一些"流浪汉"冒被嘲笑的危险, 去探索不受欢迎的领域。因为他们的努力, 人们对垃圾DNA的看法 , 特别是重复元素,于20世纪90年代初开始改变。现在,越来越多的生物学家把重复元件当作为基因组的宝库。 (注29)
尽管与普遍达尔文的假设相反,这文章得出的结论是,"重复元件不是无用的垃圾DNA,而是真核基因组的重要组成部分。"(注30)
除了重复元素,另一种柯林斯所引用的"垃圾DNA"来支持猿/人共同的祖先是"假基因"(pseudogene)。
柯林斯在《上帝的语言》中写道,人类有一个假基因(胱天蛋白酶caspase-12)是无功能的,他问道: "为什么上帝要烦劳在这个精确的位置插入这样无功能的基因呢? "(注 31)。他在他后来的书《科学与信仰的语言》(2011年)引用同样的数据, 所谓无功能的维生素C假基因。他说,"宣称人类基因组是由上帝的独立创造,而不是从一个共同的祖先进化而来,意味着神在我们的基因组中插入一个破碎的DNA片段, 这是无稽之谈。"(注32)同样,布朗大学的生物学家肯尼思.米勒援引这等假基因作为"压倒性"的证据, 因为"共同的祖先, 是唯一在同一个基因中有那么多的匹配误差的可能解释。"(注33)
但假基因真的是无功能的破碎的DNA吗?
像AREs一样,假基因的多种功能已经被发现了。(注34) 事实上,两个权威生物学家在《遗传学年度评论》(Annual Review of Genetics)写道, "已经被适当调查的假基因常常表现出功能性的作用。"(注35) 同样 2011年在RNA杂志的论文,标题为"假基因:伪功能或在健康和疾病中的重点监管",辩称假基因不应该再被假定为"垃圾"。"假基因早已被贴上'垃圾'DNA的标签,是基因组在进化过程中产生的基因失败拷贝。然而,最近的结果正在挑战这个称呼;事实上一些假基因, 似乎有调节编码蛋白质表兄弟基因的潜能。"(注36)
事实上,一项研究表明,即使是柯林斯引用的胱天蛋白酶(caspase-12)假基因(注37)也可产生一类在人体中的功能蛋白质,"CARD-only protein"。(注38,39) 这研究还表明,人类的胱天蛋白酶在某些生物途径中有相互作用, 并鼓励科学家去研究胱天蛋白酶12假基因,以了解它的功能:"因为人类的胱天蛋白酶假基因在结构上堪比ICEBERG和COP/假ICE [CARD-only蛋白质]( 译注﹕都是某些蛋白质的简称), 它们在类似的生物途径中的参与将是有趣的研究"。(注40)
虽然有许多非编码DNA我们仍然不知道它们的功能,但柯林斯是错误的,因为他简单地认为重复DNA绝大多数是无功能的是"遗传上漂流的废物 (genetic flotsam and jetsam)"或假基因是"破"的DNA。在过去的5 - 10年的基因组革命, 已发现非编码DNA元素的众多功能, 具有讽刺意味的是柯林斯本人作为前人类基因组计划带头者, 曾参加过一些这方面的研究。也许这就是为什么在柯林斯《上帝的语言》一书面世后一年, 他开始收回到他对公众推广垃圾DNA的想法, 他甚至告诉一名记者,他"已停止使用这名词。"(注41)
尽管柯林斯明显地收回这想法,他参与创立的BioLogos基金会仍继续以垃圾DNA的范式来劝导信仰团体的成员, 促使他们接纳生物进化论。(注42)。实际上以垃圾DNA来看待非编码的DNA是一个日益过时的方式,并且它在证明常见的人类与猿有共同祖先理论上的用处是值得怀疑。
染色体融合,无共同祖先
柯林斯要证明人/猿共同的祖先的第二个主要论点, 是他所宣称的人类2号染色体具有类似两个黑猩猩染色体末端对末端融合而成的结构。人类有23对染色体,而黑猩猩和其他类人猿有24对。在《上帝的语言》中柯林斯认为,这种染色体融合解释了为什么人类比猿少了一对染色体,声称"要是没有一个共同的祖先假设, 这种观察是很难理解的"。(注 43)
相反,这是很容易理解的证据,不需要假定一个共同的祖先。
就算我们接受柯林斯的声称: 人类2号染色体是融合的产品。它只是表明在我们的谱系中的某个时刻,两条染色体融合成了一条。从逻辑上来说,这方面的证据没有告诉我们任何关于我们人类的血统是否来自人与猿一个共同的祖先。它也没有告诉我们最早的人类是否像猿一样。
即使我们的祖先也曾经有过24个对染色体,他们仍然可能是基本上就像完全的现代人类。北卡罗来纳州大学夏洛特分校的人类学家乔纳森·马克斯指出,"染色体融合不是给了我们的语言,或者两足行走,或大的大脑,或艺术,或无糖口香糖。这只是一些中性变化之一,缺乏外在表现, 既不好, 也不坏。"(注44)人类染色体融合的证据充其量表明我们祖先之一经历过染色体融合事件, 这变化后来得以遗留在人群中。但是这方面的证据并非告诉我们人类是否与猿有共同的祖先。
人类染色体的融合, 没有提供特别证据, 表明人类与黑猩猩有共同的祖先。这个证据与两个立场同样兼容:(A)共同祖先, 或(B)共同设计:物种之间没有共同的祖先。
如果我们跳出达尔文框架之外,以下的情形与有共同的祖先的假设同样的可能:
1.人的血统与猿的血统是分开设计的。
2.发生过染色体融合事件。
3.这事件借着遗传瓶颈(当人口规模突然变得相当小)传遍人类。
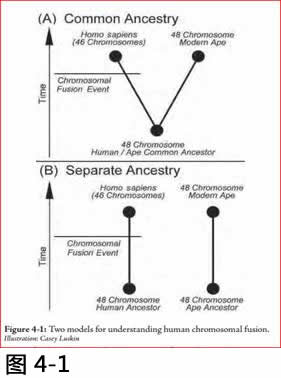
在这种情况下,证据似乎正如我们所发现的事实,没有任何与猿的共同祖先, 可由图4.1中所述的两个模式来解释。
在A模式,人类和黑猩猩有共同的祖先, 而人的血统经历了染色体融合事件, 这是柯林斯所鼔吹的标准进化模式。但是模式B是与观察数据同样兼容。在模式B 中,人类和类人猿不共享一个共同的祖先,而人的血统经历了染色体融合事件。这个模式表明,染色体融合的证据很容易被解释, 而不需要假定一个共同的祖先,没有任何共同的祖先猿。
图4.1: 两个解释染色体融合的模式
Illustration: Casey Luskin.
为了进一步说明为什么染色体融合, 并不能证明人类和类人猿有共同祖先,请考虑以下的假想情况:
试想一下, 在2050年,一个小型的孤立的人类部落经历到第二次染色体融合事件(他们保持生育性能和其他的正常生活)。我们称他们为"二次染色体融合"的人。在2100年,战争、疾病和饥荒毁灭了其余的人类。但"二次染色体融合"的人继续生存和重新充满地球,再度发现遗传和进化。最终,"二次染色体融合"的人开发技术来审视自己的染色体, 他们的科学家惊呼,"我们'二次染色体融合'的人有22对染色体,包括2对融合染色体。由于猩猩有24个对染色体, 我们必须是有48条染色体的类猿生物的后代!"
从我们的角度来看,我们可以晓得"二次染色体融合"的人的第二个染色体融合事件是最近发生的,远离人类和黑猩猩之间的共同祖先世代,并没有提供逻辑上的原因来推断人类与黑猩猩是否有共同的祖先。为什么我们应该认为在这个情况下,必须与我们的现在的一次融合染色体不同?然而许多达尔文进化论者错误地观看我们的一对染色体的融合, 正如"二次染色体融合"的人查看他们的两对融合的染色体一样。
达尔文论者可能会这样回答:"染色体融合的证据表明我们的祖先曾经有48条染色体,像黑猩猩和其他类人猿今天所有的一样。此外,我们融合后的2号染色体甚至包含类似猿染色体2a和2b的片段。共同的祖先论就已经预料到这一切的证据。"但是,达尔文论者的反驳仅仅是重申一个事实,即人类和类人猿有着高度相似的基因结构。高度的人类/黑猩猩功能基因相似性并不能说明他们有共同的祖先。在本书第一章,高杰已经阐述了为何共享的人类/黑猩猩功能和遗传相似性, 并不必然演示共同的祖先, 功能性基因的相似可能是功能性的需求或是共同设计,而不是从一个共同的祖先继承而来。事实上,正如我们已经说明过的,即使柯林斯也承认,功能基因相似性"当然本身并不能证明一个共同的祖先", 因为设计师可以"重复使用成功的设计原则。"
染色体融合的证据充其量增强我们已经知道了的理念: 黑猩猩和人类具有较高的遗传相似性。这种功能的相似性可以很容易地被解释, 是基于共同设计所实现功能需求的结果。
到目前为止我们都假设真有人类遗传史上的染色体融合事件。但是,这一论点的实际证据真是那么强吗?
生物学家肯尼斯·米勒认为, 当使用染色体融合来鼓吹人类/黑猩猩的共同血统时,"人类丢失了的染色体, 在法医检查情况下毫无疑问地得到解决。"(注45) 但事实上,染色体融合的证据不像进化论者米勒声称的那么明确。
在我们的染色体末端的端粒DNA(telomeric DNA)通常有数千个6碱基对序列TTAGGG的重复。但是,如果两条染色体融合是终端对终端:在人类2号染色体涉嫌融合点所包含的序列, 远少于它应该有的端粒DNA, 进化论生物学家丹尼尔·费尔班克斯坦言,这位置上只有158个重复序列,其中只有"44个是完美的复制品"。(注46)
此外,在《基因组研究》(Genome Research)期刊的论文中发现,我们确实有所谓融合点的端粒序列"显著地多元化"(degenerate)和"与原型(未融合前)端粒重复序列的高度分歧(diverged)"。作者惊讶于这一发现,因为融合事件据称是在人类进化史上最近才发生的事件----没有足够时间可以引致太多太新的序列,和这种戏剧性的分歧。这篇论文问道:"如果端粒重复阵列融合事件发生的时间低于约6百万年前,为什么阵列的融合点发生这么多的分歧?"(注47)所得到的结论是这样的:如果两条人的染色体融合是终端对终端,那么大量的所谓的端粒DNA已被遗失或出现乱码。
最后,端粒DNA存在于哺乳动物染色体中, 并不是特别不寻常的事,不一定代表两个古代染色体的某些融合点。进化生物学家理查德·斯滕恩伯格指出,间质性端粒序列(ITSs: Insterstitial telomeric sequences)通常散布在整个哺乳动物的基因组中,但人类2号染色体中的端粒序列, 是进化论者特别采摘引为染色体融合事件证据:
在所有已知ITSs中(许多在黑猩猩和人,以及小鼠和大鼠和牛的基因组)……。。。其中的2q13 ITS是可与进化断点或融合相关联的唯一的一个片段。我要赶紧补充指出, 其他的ITSs不与灵长类动物的染色体断点有关联。总之,要想将2q13 ITS装备作为典型的人类和黑猩猩的基因组的片段, 似乎就像采摘樱桃一样的挑选数据。大多数ITSs是不被他们描绘的DNA融合疤痕。(注48)
因此,至少有三个理由来评论这些DNA证据, 不符合染色体融合理论所预测的结果:
" 在2号染色体涉嫌融合点包含端粒DNA量比它应该有的少得多。
" 我们确实有所谓融合端粒点序列"高度多元化"和"高度分歧化",超乎我们对较近古代发生的染色体融合事件所期望的。
" 在哺乳动物基因组的中间质发现端粒DNA不是那么的不寻常,它们并不一定表明是染色体融合事件的后果。
但是关键的一点: 即使人类2号染色体是其中两个其他染色体融合的结果,这并不是人/猿共同的祖先证据。这数据充其量说明我们人类的血统中经历了染色体融合事件,但它并没有告诉我们,我们的血统是否来自一个与猿共有的祖先。
结论
近年来,遗传数据已经被提供给公众作为明确的新证据,证明人类与猩猩等动物共享一个共同的祖先。弗朗西斯·柯林斯一直积极的推广这种论调, 尤其是在信仰团体。据柯林斯所言, 是不再有任何意见分歧的空间: "基因组研究无情地导致我们人类与其他生物共享一个共同祖先的结论。"(注49)。事实上, 人/猿具有共同祖先的想法不单是不容置疑,更是"一个人类和小鼠的共同的祖先的结论几乎是不可避免。"(注50)
然而,尽管所有的柯林斯的用语,是"无情"和"不可避免的", 但事实上,他所提出基于遗传学的证据根本没有显示像他所声称的那样。
在最好的情况之下,本章中所讨论的证据,重申了一些我们已经知道的事实:人类和黑猩猩有着类似功能的DNA序列。但是,这可以通过共同的设计或共同祖先的假设, 一样可以说明。除此之外还有什么其他证据呢?不多!
正如我们前面所讨论的,柯林斯所引用的"垃圾DNA"数据, 通过新的研究, 正在逐渐被瓦解, 因为它们揭示了非编码DNA万千个特别的功能(注51),生物学家发现丰富的数据,证实非编码DNA像古代的重复元素甚至是假基因的功能---这些DNA就是弗朗西斯·柯林斯等人, 用作支持人/猿共同的祖先理论所声称为无用的"遗传上漂流的废物"。
柯林斯基于染色体融合的说法也未能成立。即使染色体融合事件确实发生过,它并不告诉我们人类的血统是否源自一个与猿分享的共同祖先。当然,我们也不能完全确定染色体融合已发生了。间质性端粒DNA并不一定表明染色体融合事件,在人类第2号染色体间隙端粒序列的"高度分化"是超越了我们对最近染色体融合事件的期望。
我支持关于自然的许多方面最好的解释, 都是智慧设计, 而不是机遇过程,但是我想指出,智慧设计与人类与其他物种共享祖先的原则不相抵触。在它的核心,智慧设计并非挑战共同的祖先的理念,而是声称生命的复杂性,通过类似随机突变和自然选择的非引导过程中出现了的立场。所以按照被指导的形式而来的人类与物种的共同祖先的理论与智慧设计兼容。
然而,智慧设计论者与达尔文进化论的支持者不同的地方,是他们没有义务接受人/猿共同的祖先作为一个大前题。他们可以跟随证据的导引来作中肯的衡量。而且这些证据并不支持弗朗西斯·柯林斯所鼓吹的结论。正如我们前面所讨论的,对于人/猿共同的祖先的遗传辩论更多是基于达尔文的假设和过时的数据, 而不是从仔细分析证据而来的结论。
注释:
1. Francis Collins, The Language of God: A Scientist Presents Evidence for Belief (New York: Free Press, 2006), 127-28.
2. See: David Klinghoffer, "Francis Collins: A Biography." Wesley J. Smith, "Collins Heads NIH," To the Point (July 30, 2009). David Klinghoffer, "Francis Collins on Abortion," BeliefNet (July 8, 2009).
3. For information about the Templeton grant to launch BioLogos, see "The Language of God: BioLogos Website and Workshop," John Templeton Foundation, accessed March 19, 2012, http://www.templeton.org/what-wefund/grants/the-language-of-god-biologos-website-and-workshop.
4. Collins, Language of God, 136-37.
5. Ibid., 138.
6. Ibid., 134.
7. Ibid., 136-37.
8. Ibid.
9. Richard Dawkins, "The Information Challenge," The Skeptic, 18 (December, 1998).
10. Richard Sternberg, "On the Roles of Repetitive DNA Elements in the Context of a Unified Genomic-Epigenetic System," Annals of the New York Academy of Sciences, 981 (2002): 154-88.
11. Ibid.
12. Sternberg, "On the Roles of Repetitive DNA Elements in the Context of a Unified Genomic-Epigenetic System," 154-88.
13. Tammy A. Morrish, Nicolas Gilbert, Jeremy S. Myers, Bethaney J. Vincent, Thomas D. Stamato, Guillermo E. Taccioli, Mark A. Batzer, and John V. Moran, "DNA repair mediated by endonuclease-independent LINE-1 retrotransposition," Nature Genetics, 31 (June, 2002): 159-65.
14. Galit Lev-Maor, Rotem Sorek, Noam Shomron, and Gil Ast, "The birth of an alternatively spliced exon: 3' splice-site selection in Alu exons," Science, 300 (May 23, 2003): 1288-91; Wojciech Makalowski, "Not junk after all," Science, 300 (May 23, 2003): 1246-47.
15. Nurit Paz-Yaacova, Erez Y. Levanonc, Eviatar Nevod, Yaron Kinare, Alon Harmelinf, Jasmine Jacob-Hirscha, Ninette Amariglioa, Eli Eisenbergg, and Gideon Rechavi, "Adenosine-to-inosine RNA editing shapes transcriptome diversity in primates," Proceedings of the National Academy of Sciences USA,
107 (July 6, 2010): 12174-79.
16. Morrish et al., "DNA repair mediated by endonuclease-independent LINE-1 retrotransposition," 159-65; Annie Tremblay, Maria Jasin, and Pierre Chartrand, "A Double-Strand Break in a Chromosomal LINE Element Can Be Repaired by Gene Conversion with Various Endogenous LINE Elements in Mouse Cells," Molecular and Cellular Biology, 20 (January, 2000): 54-60; Ulf Grawunder, Matthias Wilm, Xiantuo Wu, Peter Kulesza, Thomas E. Wilson, Matthias Mann, and Michael R. Lieber, "Activity of DNAligase IV
stimulated by complex formation with XRCC4 protein in mammalian cells," Nature, 388 (July 31, 1997): 492-95; Thomas E. Wilson, Ulf Grawunder, and Michael R. Lieber, "Yeast DNA ligase IV mediates non-homologous DNA end joining," Nature, 388 (July 31, 1997): 495-98.
17. Richard Sternberg and James A. Shapiro, "How repeated retroelements format genome function," Cytogenetic and Genome Research, 110 (2005): 108-16.
18. Jeffrey S. Han, Suzanne T. Szak, and Jef D. Boeke, "Transcriptional disruption by the L1 retrotransposon and implications for mammalian transcriptomes," Nature, 429 (May 20, 2004): 268-74; Bethany A. Janowski, Kenneth E. Huffman, Jacob C. Schwartz, Rosalyn Ram, Daniel Hardy, DavidS. Shames, John D. Minna, and David R. Corey, "Inhibiting gene expression at transcription start sites in chromosomal DNA with antigene RNAs," Nature Chemical Biology, 1 (September, 2005): 216-22; J. A. Goodrich, and J. F. Kugel, "Non-coding-RNA regulators of RNA polymerase II transcription," Nature Reviews Molecular and Cell Biology, 7 (August, 2006): 612-16; L.C. Li, S. T. Okino, H. Zhao, H., D. Pookot, R. F. Place, S. Urakami, H. Enokida, and R. Dahiya, "Small dsRNAs induce transcriptional activation in human cells," Proceedings of the National Academy of Sciences USA, 103 (November 14, 2006): 17337-42; A. Pagano, M. Castelnuovo, F. Tortelli, R. Ferrari, G. Dieci, and R. Cancedda, "New small nuclear RNA gene-like transcriptional units as sources of regulatory transcripts," PLoS Genetics, 3(February, 2007): e1; L. N. van de Lagemaat, J. R. Landry, D. L. Mager, and P. Medstrand, "Transposable elements in mammals promote regulatory variation and diversification of genes with specialized functions," Trends in Genetics, 19 (October, 2003): 530-36; S. R. Donnelly, T. E. Hawkins, and S. E. Moss, "A Conserved nuclear element with a role in mammalian gene regulation," Human Molecular Genetics, 8 (1999): 1723-28; C. A. Dunn, P. Medstrand, and D. L. Mager, "An endogenous retroviral long terminal repeat is the dominant promoter for human B1,3-galactosyltransferase 5 in thecolon," Proceedings of the National Academy of Sciences USA, 100 (October 28, 2003):12841-46; B. Burgess-Beusse, C. Farrell, M. Gaszner, M. Litt, V. Mutskov, F. Recillas-Targa, M. Simpson, A. West, and G. Felsenfeld, "The insulation of genes from external enhancers and silencing chromatin," Proceedingsof the National Academy of Sciences USA, 99 (December 10, 2002): 16433-37; P. Medstrand, Josette-Renée Landry, and D. L. Mager, "Long Terminal Repeats Are Used as Alternative Promoters for the Endothelin B Receptor and Apolipoprotein C-I Genes in Humans," Journal of Biological Chemistry, 276 (January 19, 2001): 1896-1903; L. Mari?o-Ramíreza, K.C.Lewisb, D. Landsmana, and I.K. Jordan, "Transposable elements donate lineage-specific regulatory sequences to host genomes," Cytogenetic and Genome Research, 110 (2005):333-41.
19. S. Henikoff, K. Ahmad, and H. S. Malik "The Centromere Paradox: Stable Inheritance with Rapidly Evolving DNA," Science, 293 (August 10, 2001):1098-1102; C. Bell, A. G. West, and G. Felsenfeld, "Insulators and Boundaries: Versatile Regulatory Elements in the Eukaryotic Genome," Science, 291
(January 19, 2001): 447-50; M.-L. Pardue & P. G. DeBaryshe, "Drosophila telomeres: two transposable elements with important roles in chromosomes," Genetica, 107 (1999): 189-96; S. Henikoff, "Heterochromatin function in complex genomes," Biochimica et Biophysica Acta, 1470 (February, 2000):
O1-O8; L. M.Figueiredo, L. H. Freitas-Junior, E. Bottius, Jean-Christophe Olivo-Marin, and A. Scherf, "A central role for Plasmodium falciparum subtelomeric regions in spatial positioning and telomere length regulation," The EMBO Journal, 21 (2002): 815-24; Mary G. Schueler, Anne W. Higgins, M. Katharine Rudd, Karen Gustashaw, and Huntington F. Willard, "Genomic and Genetic Definition of a Functional Human Centromere," Science, 294 (October 5, 2001): 109-15.
20. Ling-Ling Chen, Joshua N. DeCerbo and Gordon G. Carmichael, "Alu element-mediated gene silencing," The EMBO Journal 27 (2008): 1694-1705; Jerzy Jurka, "Evolutionary impact of human Alu repetitive elements," Current Opinion in Genetics & Development, 14 (2004): 603-8; G. Lev-Maor et al.
"The birth of an alternatively spliced exon: 3' splice-site selection in Alu exons," 1288-91; E. Kondo-Iida, K. Kobayashi, M. Watanabe, J. Sasaki, T. Kumagai, H. Koide, K. Saito, M. Osawa, Y. Nakamura, and T. Toda, "Novel mutations and genotype-phenotype relationships in 107 families with Fukuyamatype
congenital muscular dystrophy (FCMD)," Human Molecular Genetics, 8 (1999): 2303-09; John S. Mattick and Igor V. Makunin, "Non-coding RNA," Human Molecular Genetics, 15 (2006): R17-R29.
21. M. Mura, P. Murcia, M. Caporale, T. E. Spencer, K. Nagashima, A. Rein, and M. Palmarini, "Late viral interference induced by transdominant Gag of an endogenous retrovirus," Proceedings of the National Academy of Sciences USA, 101 (July 27, 2004): 11117-22; M. Kandouz, A. Bier, G. D Carystinos, M. A Alaoui-Jamali, and G. Batist, "Connexin43 pseudogene is expressed in tumor cells and inhibits growth," Oncogene, 23 (2004): 4763-70.
22. K. A. Dunlap, M. Palmarini, M. Varela, R. C. Burghardt, K. Hayashi, J. L. Farmer, and T. E. Spencer, "Endogenous retroviruses regulate periimplantation placental growth and differentiation," Proceedings of the National Academy of Sciences USA, 103 (September 26, 2006): 14390-95; L. Hyslop, M. Stojkovic, L. Armstrong, T. Walter, P. Stojkovic, S. Przyborski, M. Herbert, A. Murdoch, T. Strachan, and M. Lakoa, Downregulation of NANOG Induces Differentiation of Human Embryonic Stem Cells to Extraembryonic
Lineages," Stem Cells, 23 (2005): 1035-43; E. Peaston, A. V. Evsikov, J. H. Graber, W. N. de Vries, A. E. Holbrook, D. Solter, and B. B. Knowles, "Retrotransposons Regulate Host Genes in Mouse Oocytes and Preimplantation Embryos," Developmental Cell, 7 (October, 2004): 597-606.
23. Sternberg Shapiro, "How Repeated Retroelements format genome function," 108-16.
24. Rick Weiss, "Intricate Toiling Found In Nooks of DNA Once Believed to Stand Idle," Washington Post (June 14, 2007), accessed March 6, 2012, http://www.washingtonpost.com/wp-dyn/content/article/2007/06/13/AR2007061302466_pf.html.
25. Erika Check Hayden, "Human Genome at Ten: Life is Complicated," Nature, 464 (April 1, 2010): 664-67.
26. Philipp Kapranov, Aarron T. Willingham, and Thomas R. Gingeras, "Genome-wide transcription and the implications for genomic organization," Nature Reviews Genetics, 8 (June, 2007): 413-23.
27. Paulo P. Amaral, Marcel E. Dinger, Tim R. Mercer, and John S. Mattick, "The Eukaryotic Genome as an RNA Machine," Science, 319 (March 28, 2008): 1787-89.
28. Ibid.
29. Makalowski, "Not Junk After All," 1246-47.
30. Ibid.
31. Collins, The Language of God, pg. 139.
32. Karl Giberson and Francis Collins, The Language of Science and Faith: Straight Answers to Genuine Questions (Downers Grove, IL: InterVarsity Press, 2011), 43.
33. Private correspondence with Dr. Miller.
34. See for example D. Zheng and M. B. Gerstein, "The ambiguous boundary between genes and pseudogenes: the dead rise up, or do they?," Trends in Genetics, 23 (May, 2007): 219-24; S. Hirotsune et al., "An expressed pseudogene regulates the messenger-RNA stability of its homologous coding gene," Nature, 423 (May 1, 2003): 91-96; O. H. Tam et al., "Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes," Nature, 453 (May 22, 2008): 534-38; D. Pain et al., Multiple Retropseudogenes from Pluripotent Cell-specific Gene Expression Indicates a Potential Signature for
Novel Gene Identification," The Journal of Biological Chemistry, 280 (February 25, 2005):6265-68; J. Zhang et al., "NANOGP8 is a retrogene expressed in cancers," FEBS Journal, 273 (2006): 1723-30.
35. Evgeniy S. Balakirev and Francisco J. Ayala, "Pseudogenes, Are They 'Junk' or Functional DNA?," Annual Review of Genetics, 37 (2003): 123-51.
36. Ryan Charles Pink, Kate Wicks, Daniel Paul Caley, Emma Kathleen Punch, Laura Jacobs, and David Paul Francisco Carter, "Pseudogenes: Pseudo-functional or key regulators in health and disease?," RNA, 17 (2011): 792-98.
37. Collins acknowledges that the caspase-12 gene produces a full-fledged protein in chimps, so this is not a case where humans share a non-functional stretch of DNA with another species. In fact, the gene is not always a pseudogene in humans. According to a paper in The American Journal of Human Genetics,28% of people in sub-Saharan Africa have a functioning copy of the caspase-12 gene, as do lower percentages in some other human populations. Collins ignores the obvious possibility that caspase-12 was originally designed to produce a functional protein in humans but was rendered noncoding bya mutation in some human populations at some point the recent past. See Yali Xue, Allan Daly, BryndisYngvadottir, Mengning Liu, Graham Coop, Yuseob Kim, Pardis Sabeti, Yuan Chen, Jim Stalker, Elizabeth Huckle, John Burton, Steven Leonard, Jane Rogers, and Chris Tyler-Smith, "Spread of an Inactive Form of Caspase-12 in Humans Is Due to Recent Positive Selection," The American Journal of Human Genetics, 78 (April, 2006): 659-70.
38. M. Lamkanfi, M. Kalai, and P. Vandenabeele, "Caspase-12: an overview," Cell Death and Differentiation, 11: (2004)365-68.
39. Sug Hyung Lee, Christian Stehlik, and John C. Reed, "COP, a Caspase Recruitment Domain-containing Protein and Inhibitor of Caspase-1 Activation Processing," The Journal of Biological Chemistry, 276 (September 14, 2001): 34495-500.
40. Lamkanfi, Kalai, and Vandenabeele, "Caspase-12: an overview," 365-68.
41. Collins, quoted in Catherine Shaffer, "One Scientist's Junk Is a Creationist's Treasure," Wired Magazine Blog (June 13, 2007), accessed March 6, 2012.
42. See discussion in Jonathan Wells, The Myth of Junk DNA (Seattle: Discovery Institute Press, 2011), 98-100.
43. Collins, The Language of God, 138.
44. Jonathan Marks, What it means to be 98% Chimpanzee: Apes, People, and their Genes (Los Angeles: University of California Press, 2003), 39.
45. Kenneth R. Miller, Only a Theory: Evolution and the Battle for America's Soul (New York: Viking, 2008), 107.
46. Daniel Fairbanks, Relics of Eden: The Powerful Evidence of Evolution in Human DNA (Amherst, NY: Prometheus, 2007), 27.
47. Yuxin Fan, Elena Linardopoulou, Cynthia Friedman, Eleanor Williams, and Barbara J. Trask, "Genomic Structure and Evolution of the Ancestral Chromosome Fusion Site in 2q13-2q14.1 and Paralogous Regions on Other Human Chromosomes," Genome Research, 12 (2002): 1651-62.
48. Richard Sternberg, "Guy Walks Into a Bar and Thinks He's a Chimpanzee:The Unbearable Lightness of Chimp-Human Genome Similarity," Evolution News & Views (May 14, 2009), accessed March 6, 2012.http://www.evolutionnews.org/2009/05/guy_walks_into_a_bar_and_think020401.html (internal citations removed).
49. Collins, The Language of God, 133-34.
50. Ibid., 136-37.
51. For an in-depth discussion of these studies, see Wells, The Myth of Junk DNA.